| المجلد 3 , العدد 9 , رمضان 1426 - تشرين الأول (أكتوبر) 2005 |
| |
| نظام رُقَاقات البروتين البيولوجية |
| Protein Biochips System |
| د. محمد كامل كبه وار |
| Kebbewar M.K. |
| المقدمة Introduction |
لم يكن هنالك طرق كافية لفهم الآليات المعقدة والتي تربط بين العمليات البيولوجية والأمراض، لذلك طور العلماء طرقاً جديدة في نظام علم الأحياء تعتمد على تنظيمات وسبل معقدة. تعتمد هذه الأنظمة على تحديد بروفيل التعبير الجيني Gene Expression وتحليل الـ DNA، وقد أعطت هذه المقاربة فرصة لمعرفة المجين (الجينوم genome) والتركيز على دراسة التغيرات التي تتناول مجموعة معينة من الجينات.
من التقانات الحديثة التي تم تطويرها في هذا المجال تقانة تدعى مصفوفات مكروية Microarrays وهي تقانة أحدثت ثورة في مجال دراسات تحليل التعبير الجيني وفي إمكانية دراسة التغيرات المتواصلة في هذا التعبير لآلاف الجينات، ولكن المشكلة كانت أن التبدلات التي يمكن رؤيتها على مستوى البروتين يمكن أن لا تظهر على مستوى تبدل الـ RNA والعكس صحيح، لذا كان لابد من إيجاد ما يسمى برُقَاقات البروتين البيولوجية Protein Biochips والتي تشير عادة إلى مصفوفات البروتين المكرويةProtein Microarrays وهي عبارة عن ترتيب منظم لعدة بروتينات كالأجسام المضادة Antibodies ومعروفة النظائر على رقاقة Chip.
تعمل هذه البروتينات كعامل ارتباط للبروتينات المختارة في العينة، والتي يمكن كشفها فيما بعد (أي البروتينات في العينة المدروسة) عن طريق التوسيم Labeling بشكل مباشر وإظهارها بخاصية الفلورة (كما في حالة رُقَاقات الأجسام المضادة Antibody Chips بوساطة إضافة طبقة من الأجسام المضادة الموسومة كمبدأ الشطيرة Sandwich (الشكل 1).
يمكن باستخدام تقانة مصفوفة الأضداد المكروية Antibodies microarray تحليل الآلاف من البروتينات في قطرة من عينة مثل مصل الدم، كما وظفت هذه التقانة لخدمة الأبحاث الطبية والبيولوجية جاعلةً إياها أسهل لدراسة التفاعلات الحيوية الجزيئية أو لكشف نوع معين من البروتينات ضمن مزيج معقد (الشكل 2).
تغلّبَ نظـام رُقَاقات البروتين البيولوجية Protein biochips على التحديات التي لم يتم تجاوزها باستخدام نظام رُقَاقات الدنا DNA Chips إذ أنه من الممكن أن يكون هناك 1 مليون بروتين بشري مختلف مقارنة مع 3000 أو أكثر من الجينات، بالإضافة إلى أن نظام Protein biochips قادر على كشف كميات قليلة جداً من البروتين وذلك دون الحاجة إلى استخدام تقانة المضاعفة كاستخدام التفاعل السلسلي للبوليميراز PCR.
يعد نظام رُقَاقات البروتين البيولوجية Protein biochips مفيداً في جميع المجالات تقريباً وفي تطوير الأدوية وفي المقام الأول لتحديد الأمراض وإمكانية اختبار نوعية وانتقائية عقار ما وتقدير آلية عمله، وفي الدراسات التحليلية (السريرية) وفي مجالات طبية وتشخيصية مرضية أخرى.
يكفي استخدام 75 مكرولتر من العينة (مصل مثلاً) من أجل كل مصفوفة Array لتطبيق هذه التقانة والكشف عن البروتينات الهدف، كما أن جميع خطوات العمل تتم بشكل آلي ومباشر وذلك بالاستعانة بالبرنامج الخاص بالكمبيوتر. يعمل هذا النظام مع أجسام مضادة نموذجية (بروتين - Antibodies) أو حسب تفاعل (بروتين – بروتين) حيث يكون البروتين الثاني موسوماً labeled بشكل مباشر، وكما ذكر سابقاً يمكن لهذه التقانة الكشف حتى عن الكميات القليلة من البروتين في العينة المدروسة وذلك بفضل خاصية وجود ماسح ليزري يقيس شدة الفلورة.
يُعد نظام رُقَاقات البروتين البيولوجية Protein biochips مناسباً من أجل المصفوفات المكروية Microarrays للبروتين والأجسام المضاد والمستضدات Antigens والببتيدات والـ DNA والجزئيات الصغيرة small molecules والسكريات Carbohydrates والشحوم Lipids إذا كان بالإمكان تحضيرها كمعلق.
إذا كان هدف رُقَاقات البروتين البيولوجية Protein biochips المصممة بشكل رئيسي هو الكشف عن وجود أو غياب بروتين معين في العينات البيولوجية المفحوصة فإن نظام رُقَاقات التآثر Interaction chips يمكن أن يطبق لمعرفة طبيعة البروتينات ووظيفتها بوساطة قياس الوقت الحقيقي Real-Time measurement لنوعية وحركية ومدى الصلة (الألفة) بين البروتينات واللجائن ligands، بين بروتين وآخر، أو بين بروتينات متعددة. إن الكفاءة العالية لهذه التقانة تسمح بتحليل تراكيز متعددة ومتكررة لكل عينة هدف على رقاقة chip واحدة وخلال تجربة واحدة (الشكل 3).
سيصبح استخدام نظام رُقَاقات البروتين البيولوجية Protein biochips في السنوات القليلة القادمة مستخدماً بشكل كبير ومألوف، ولعل الأمر الأكثر أهمية من حيث استخدام هذه التقانات هو الحصول على الدعم والتدريب الخاصين. إن نظام Protein biochips سيصبح أداة شائعة في كل بحث وفي كل مخبر تشخيصي حول العالم.
|
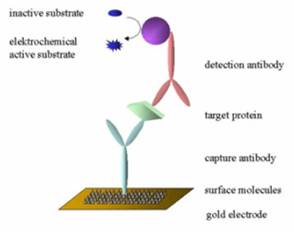
الشكل 1: مبدأ الكشف في نظام رُقَاقات البروتين البيولوجية
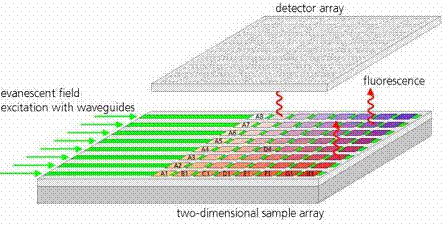
الشكل 2: مبدأ الكشف بطريقة المصفوفة المكروية.
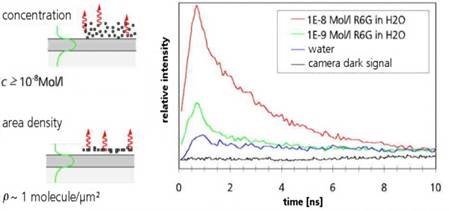
الشكل 3: مبدأ تحليل تراكيز متعددة في البروتينات باستخدام تقانة رُقَاقات البروتين البيولوجية |
| المراجع Reference |
1-Lockhart DJ. and Winzeler EA.
Genomics, gene expression and DNA arrays.
Nature, 405: 827-836, 2000.
2-Lockhart DJ; Dong H; Byrne MC; Follettie MT; Gallo MV; Chee MS; Mittmann M; Wang C; Kobayashi M; Horton H. and Brown EL.
Expression monitoring by hybridization to high-density oligonucleotide arrays.
Nature Biotechnol, 14: 1615-1680, 1996.
3-Duggan DJ; Bittner M; Chen Y; Meltzer P. and Trent JM.
Expression profiling using cDNA microarrays.
Nature Genetics, 21: 10-14, 1999.
4-Young RA.
Biomedical discovery with DNA arrays.
Cell, 102: 9-15, 2000.
5-Golub TR; Slonim DK; Tamayo T; Huard C; Gaasenbeek M; Mesirov JP; Coller H; Loh ML; Downing JR; Caligiuri MA; Bloomfield CD. and Lander ES.
Molecular classification of cancer: class discovery and class prediction by gene expression.
Science, 286: 531-537, 1999.
6-van't Veer LJ; Dai H; van de Vijver MJ; He YD; Hart AAM; Mao M; Peterse HL; van der Kooy K; Marton MJ; Witteveen AT; Schreiber GJ; Kerkoven RM; Roberts C; Linsley PS; Bernards R. and Friend SH.
Gene expression profiling predicts clinical outcome of breast cancer.
Nature, 415, 530-536, 2002.
7-Pomeroy SL; Tamayo P; Gaasenbeek M; Sturla LM; Angelo M; McLaughlin ME; Kim Jr; Goumnerova LC; Black PM; Lau C; Allen JC; Zagzag D; Olson JM; Curran T; Wetmore C; Biegel JA; Poggio T; Mukherjee S; Rifkin R; Califano A; Stolovitzky G; Louis DN; Mesirov JP; Lander ES. and Golub TR.
Prediction of central nervous system embryonal tumour outcome based on gene expression.
Nature, 415: 436-442, 2002.
8-Marton MJ; DeRisi JL; Bennett HA; lyer VR; Meyer MR; Roberts C; Stoughton R; Burchard J; Slade D; Dai H; Bassett DE; Hartwell LH; Brown PO. and Friend SH.
Drug target validation and identification of secondary drug target effects using DNA microarrays.
Not. Med., 4: 1293-1301, 1998.
9-Tavazoie S; Hughes JD; Campbell MJ; Cho RJ. And Church GM.
Systematic determination of genetic network architecture.
Nature Genetics, 22: 281-285, 1999.
10-Waring JF; Ciurlionis R; Jolly RA; Heindel M. and Ulrich RG.
Microarray analysis of hepa- totoxins in vitro reveals a correlation between gene expression profiles and mechanism! of toxicity.
Toxicol Letters, 120: 359-368, 2001.
11-TheilhaberJ; Connolly T; Roman-Roman S; Bushnell S; Jackson A; Call K; Garcia T. and Baron R.
Finding genes in the C2C12 osteogenic pathway by k-nearest-neighbor classification of expression data.
Genome Res., 1: 165-176, 2002.
12-DeRisi JL; lyer VR. and Brown PO.
Exploring the metabolic and genetic control of gene expression on a genomic scale.
Science, 278: 680-686, 1997.
13-Iyer VR; Eisen MB; Ross DT; Schuler G; Moore T; Lee JCF; Trent JM; Staudt LM; Hudson J; Boguski MS; Lashkari D; Shalon D; Botstein D. and Brown PO.
The transcriptional program in the response of human fibroblasts to serum.
Science, 283: 83-87, 1999.
14-Hughes TR; Marton MJ; Jones AR; Roberts CJ; Stoughton R; Armour CD; Bennett HA; Coffey E; Dai H; He YD; Kidd MJ; King AM; Meyer MR; SladeD; Lum PY; Stepaniants SB; Shoemaker DD; Gachotte D; Chakraburtty K; Simon J; Bard M. and Friend SH.
Functional discovery via a compendium of expression profiles.
Cell, 102: 109-126, 2000.
15-Mffliken GA and Johnson DE.
Analysis of Messy Data, Volume 1: Designed Experiments.
Wadsworth, Inc. Belmont, California; 1984.
16-Kerr MK. and Churchill G.A.
Experimental design for gene expression microarrays.
Biostatistics, 2: 183-201, 2001.
17-Gaasterland T and Bekiranov S.
Making the most of micro array data.
Nature Genetics, 24: 204-206, 2002.
18-Zolman JF.
Biostatistics: Experimental design and statistical inference.
Oxford University Press, Oxford; 1993.
19-Hwang D; Schmitt WA. and Stephanopoulos G.
Determination of minimum sample size and discriminatory expression patterns in microarray data.
Bioinjormatics, 18: 1184-1193, 2002.
20-Kerr MK; Martin M. and Churchill GA.
Analysis of variance for gene expression microarray data.
J. Compo. Biol., 7: 819-837, 2000.
21-Kane MD; Jatkoe TA; Stumpf CR; Lu J; Thomas JD. and Madore SJ.
Assessment of the sensitivity and specificity of oligonucleotide (50 mer) microarrays.
Nuc. Acids. Res., 28: 4552-4557, 2000.
22-Brody JP; Williams BA; Wold BJ. and Quake SR.
Significance and statistical errors in the analysis of DNA microarray data.
Proc. Not. Acad. Sci., 99: 12975-12978, 2002.
23-Hughes TR; Mao M; Jones AR; Burchard J; Marton MJ; Shannon KW; Lefkowitz SM; Ziman M; Schelter JM; Meyer MR; Kobayashi S; Davis C; Dai H; He YD; Stephaniants SB; Cavet G; Walker WL; West A; Coffey E; Shoemaker DD; Stoughton R; Blanchard AP; Friend SH. and Linsley PS.
Expression profiling using microarrays fabricated by an ink-jet oligonucleotide synthesizer.
Nat. Biotech., 19: 342-347, 2001.
24-Ramakrishnan R; Dorris DR; Lublinsky A; Nguyen A; Domanus M; Prokhorova A; Gieser L; Touma E; Lockner R; Tata M; Shippy R; Sendera T. and Mazumder A.
An assessment of Motorola CodelinkTM microarray performance for gene expression profiling applications.
Nucleic Acids Res. 30: 30, 2002.
25-Yuen T; Wurmbach E; Pfeffer RL; Ebersole BJ. and Sealfon SC.
Accuracy and calibration of commercial oligonucleotide and custom cDNA microarrays.
Nucleic Acids Res., 30: 48, 2002.
|
| |
| |
| المجلد 3 , العدد 9 , رمضان 1426 - تشرين الأول (أكتوبر) 2005 |
|
|
|
