| المجلد 6 ,
العدد 2
, رجب 1432 - تموز (يوليو) 2011 |
| |
| التطبيقات السريرية المتقدمة لتكنولوجيا الـ PCR في الوراثيات الجزيئية والسرطان |
| Advanced Clinical Applications of PCR Technology
in the Molecular Genetics & Cancer
|
| د. كامل كبه وار |
| (مقالة مرجَعَية Review Article) |
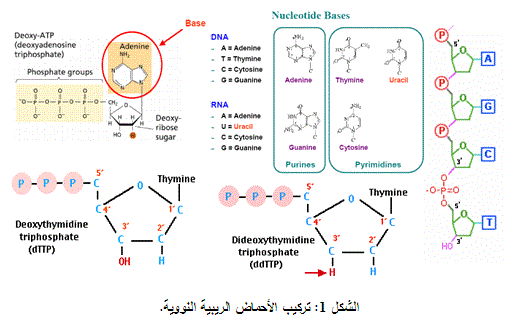
مع دخولنا الألفية الجديدة سيكون لتطبيقات تقانات الدنا (DNA) (موضح تركيبه في الشكل 1) في مجال الوراثيات الجزيئية مردود عظيم في خدمة الإنسانية، لا سيما مع ظهور تطبيقات المُعالَجَةُ الجِينيَّةgene therapy على الإنسان، وخاصةً في مجال تحري الطفرات المسببة للأمراض، والذي طُبق عملياً حتى قبل الولادة، وتنفيذ التحري الوراثي أيضاً على الأفراد قبل ظهور الأعراض بهدف كشف العوامل الممرضة المعدية بحساسية عالية، لا سيما الفيروسات وتعيين نمطها الجيني، مما يسهل تحديد مسار المعالجة بنجاعة.
حالياً نحى العلم الجزيئي باتجاه تحري الأمراض الناجمة عن طفرات جينات عدة، كما هو في السرطانات، وكذلك تعيين هوية أعداد متزايدة من الأمراض الوراثية الناجمة عن تعدد الأشكال النُوكْلِيُوتيدية المفردة (SNPs) single nucleotide polymorphisms.
سنعرض في هذه المراجعة لأهم التطبيقات الحديثة للطرق الوراثية الجزيئية:
1- التعرف على الرَوامِز الوراثية- تعيين تسلسل الدنا DNA sequencing:
تتمثل الإجابة في أربع نقاط:
1. لإيجاد الجينات بطريقة أسرع وأسهل؛
2. لمعرفة عمل هذه الجينات ومدى ارتباطها مع بعضها البعض وتداخلها معاً؛
3. بما أن مناطق الجينات تمثل أقل من 25% من حجم المجين البشري، فإن معرفة الروامز كاملة ستمكننا من دراسة المناطق المحيطة بالجينات، مما قد يسهل معرفة سبل تنشيط الجينات وإيقاف عملها؛
4. لمساعدة الأطباء في تشخيص أكثر دقة للتركيبة الوراثية للمريض.
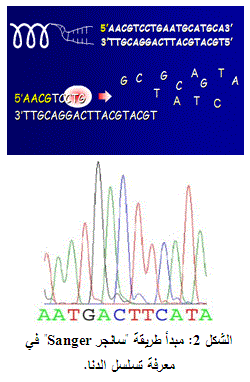
منذ ما يزيد على عشرين عاماً جرى نشر طريقتين للتعرف على الروامز الوراثية للإنسان: تدعى إحداهما طريقة سانجرSanger (الشكل 2)، نسبة إلى فريدريك سانجر، وتعتمد على تركيب سلاسل مفردة للشريط الجيني تنتهي أطرافها بقواعد نوكليوتيدية ثنائية منزوعة الأكسجين (ddATPs, ddTTPs, ddCTPs, ddGTPs)، بما لا يتيح لها الاستمرار في متابعة النسخ. وتدعى الأخرى بطريقة ماكسام جيلبرت، والتي تتضمن تقطيعاً كيميائياً لقطع الشريط الوراثي الموسوم. إن كلتا الطريقتين تتطلب أربعة تفاعلات تُقرأ لاحقا بشكل مستقل، يشير كل منها إلى أساس آزوتي، كما تتطلب هلامة البولي أكريلاميد العالي الدقة للفصل بين الشرائط الوراثية على مستوى الاختلاف في نوكليوتيد واحد. كانت طريقة سانجر أسهل وأسرع وأصبحت الطريقة الأكثر تطبيقاً.
لقد مكنت اكتشافات وتطور علم الحاسوب من أتمتة تحليل الرحلان في طريقة سانجر بسهولة ودقة أكثر، كما مكن تطور علم الكيمياء الحيوية واستخدام طريقة وسم الأسس النوكليوتيدية بألوان مختلفة من خلط الأسس الموسومة والمنزوعة الأكسجين مع بعضها، في تفاعل، وتمرير نواتج التفاعل على قارئ الكتروني قادر على الفصل بين ألوان القواعد النوكليوتيدية المختلفة.
من الطبيعي أن كشف الترميز الوراثي لا يجري باختبار واحد وإنما يحتاج إلى جهود كبيرة، كون الطرق المتاحة حالياً لا تسمح بالكشف إلا عن قطعة صغيرة جداً من الروامز الكلية. وبالتالي لا بد للباحثين من تقطيع الشريط الوراثي إلى قطع صغيرة والتعامل مع كل منها على حده (نسيلة بنسيلة clone by clone)، وبعدها تجمع النتائج وتصدر كروامز متصلة، باستخدام أجهزة حاسوب متطورة.
هنالك طريقة أخرى لفك الترميز تدعى رجْم المجين بكامله Whole-genome shotgun، تعتمد على تقطيع الشريط الوراثي إلى قطع صغيرة جداً ويجري تحليل القطع جميعها عشوائياً، ومن ثم تجمع النتائج باستخدام أجهزة حاسوب متطورة.
الطريقة الأولى أكثر مصداقية ولكنها بطيئة، فهي تستهلك وقتاً وجهداً كبيرين، بينما الطريقة الثانية سريعة ولكن تكمن صعوبتها في مرحلة تجميع الترميز.
هنالك إمكان أيضا لفك الترميز الوراثي مباشرةً لأي من نواتج تفاعل سلسلة البوليميراز والمعروف باسم (PCR) polymerase chain reaction. وهي طريقة مبتكرة لاكتشاف الطفرات مباشرة مقارنة مع الطريقة القديمة التي تعتمد على الربط ببلازميدة النقل إلى الجراثيم.
|
|
| تعتمد هذه الطريقة على استخدام أحد المَشْارع primer مباشرة مع قطعة الـ DNA الناتجة عن تفاعل الـ PCR والتي تعد كقالب في تفاعل السَلْسَلة. وغالباً ما تستخدم هذه الطريقة للتعرف على القطع غير معروفة التتالي النوكليوتيدي مسبقاً. ينصح في هذه الطريقة بتنقية نواتج تفاعل الـ PCR من الأملاح والمشارع والنوكليوتيدات الزائدة، كما يجب أحياناً اللجوء إلى قص قطعة الـ DNA من الهلامة لتنقيتها من قطع الـ DNA الأخرى والتي نتجت من تفاعل الـ PCR. |
2- تفاعل سلسلة البوليميراز (PCR) Polymerase chain Reaction
ساهمت طريقة الـ PCR منذ اكتشافها بإحداث ثورة في البحوث التطبيقية، ففي عام 1993 مُنح Mullis جائزة نوبل في الكيمياء لتطويره هذه الطريقة، ولقد أدى إدخال إنزيم البوليميراز من جرثومة الينابيع الحارة Thermus Aquaticus وتطوير أجهزة مؤتمتة، تؤدي تحاليل دقيقة إلى جعل تفاعل الـ PCR نوعياً جداً. ولاحقاً أدى استخدام تقانة الـ PCR (الشكل 3) في تضخيم مكان معين من المجين البشري إلى إدخالها في طرق لها استخدامات واسعة في مجال البحث العلمي تتضمن: تشخيص الأمراض الوراثية البشرية، تعيين الهوية البيولوجية، تشخيص الأمراض المعدية وتدبيرها، تشخيص الأمراض الدموية وتحديد مراحلها، واختبارات تقصي اختطار الإصابة بالسرطان.
على الرغم من أن العديد وربما غالبية تقانات تحري الطفرات تعتمد على طريقة التفاعل سلسلة البوليميراز للـ DNA (PCR)، إلا أن الغالبية العظمى من الطرق، وحتى الـ PCR، لا يمكن أن تكشف الطفرة الفعلية (كتتالي نوكليوتيدات). على كل حالٍ هنالك طرق تقوم على تعديل الـ PCR يمكن اعتمادها كنظام تحري طفرات أولي، حيث يختلف تصميم نظم التحليل باختلاف درجة التغايرية Heterogeneity الأليلية أو الاختلاف في عدد الطفرات للجين الواحدة. فبالنسبة للاضطرابات ذات التغايرية الأليلية المرتفعة جرى تحليل عدد محدود من الطفرات، مثل الطفرة المسببة للتليف الكيسي cyctic fibrosis. أما بالنسبة للاضطرابات ذات المجال الواسع من الطفرات، مثل حثل دوشن/ بيكر المتعدد، كانت هناك حاجة لطريقة تحري هذه الطفرات، وتعد طرق التحري مناسبة لتحليل جينات مرضية جديدة. ونذكر من هذه الطرق:
1. Real-Time PCR (Klein, 2002 وZimmermann et al., 2002).
2. MLPA: Multiple Ligation –Dependent Probe Amplification تضخيم المسبار المعتمد على الربط المتعدد (Schouten et al., 2002).
3. DNA- Methylation مَثْيَلَةٌ الدنا DNA (Brind, 1986).
4. PCR sequencing سَلْسَلة Stalko) PCR et al.,1998 ,Ansonge et al 1987 ).
5. Microsatellite الساتل الصغري (Goldstein et al., 1995).
يجب الأخذ بالحسبان أن طريقة الرحلان الكهربائي يمكن أن يُحتاج إليها لفصل قطع الـDNA التي يجري تضخيمها.
|
3- تفاعل سلسلة البوليميراز بالزمن الحقيقيّ
Real-Time Polymerase Chain Reaction (RT-PCR)
هو طريقة كميةٌ جزيئية تعمل على تحويل الحمض الريبي النووي المِرْسال mRNA أو الـ RNA غير المُرَمِّزْ Non-coding إلى دنا متمم cDNA، وذلك باستخدام إنزيم الناسِخَةِ العَكْسِيَّةِ Reverse Transpcriptase. ثم يجري تضخيم موقعٍ محددٍ أو أكثر من الدنا وذلك باستخدام إنزيم بوليميراز Taq-DNA Polymerase. ويظهر القياس الكميّ للدنا المُضَخَّمِ مباشرةً مع نهاية كلِّ دورة من التفاعل، وذلك بدون الحاجة للانتظار حتى نهاية التفاعل، ومن هنا جاء اسم الطريقة.
|
 |
تُنظِّمُ الخلايا كلاًّ من التعبير الجيني Gene Expression والمنتج البروتينيّ الناجم عن هذا التعبير. ولذلك تعد تقانة الـ RT-PCR وسيلةً عمليةً للتحري الكميِّ، وذلك عبر قياس الكميات الضئيلة جداً من الـ mRNA. كما يمكن استخدام الطريقة في كشف وتقدير وجود تتالي معين من الدنا DNA في عينات اختبارية.
تقوم الطريقة على استخدام أصبغة متفلورة، حيث يرتبط الصباغ المتفلور بشكلٍ نوعيٍّ مع كامل الدنا مضاعف السلسلة dsDNA معطياً فلورة بنسبةٍ محددة. وفي تفاعل الـ RT-PCR، فإنَّ تضخيم منطقةٍ محددةٍ من عينة الدنا سوف يزيد من شدة الفلورة، وذلك بسبب ازدياد ارتباط الأصبغة المتفلورة، وتُقاس الفلورة مع نهاية كلِّ دورة تفاعلية، مما يسمح بالقياس الكميّ للدنا المُضَخَّمْ في منطقة ذات تتالي معين من النوكليوتيدات.
|
خطوات عمل طريقة الـ RT-PCR:
يُسْتَعْمَلُ مشرعان Primers يحددان ويحصران منطقةً من الـدنا يُرادُ تضخيمها، ومسباران، الأول مسبارٌ متفلورٌ مُخبِر Fluorescent Reporter والثاني مسبار مُخَمِّدٌ Fluorescent Quencher، ويكونان عادةً قريبين جداً من بعضهما. تؤدي الزيادة في تضخيم الدنا DNA مع كل دورةٍ لمنطقة المسبار المخبر إلى زيادة نسبيةٍ في الفلورة، ويعود ذلك إلى تحرر المسبار المخبر عن المسبار المخمد.
ويجري ذلك تبعاً للخطوات التالية:
1. تُحضَّرُ العينة التفاعلية للـ RT-PCR ويُضاف المسباران المتفلوران.
2. يبدأ تفاعل الـ RT-PCR عبر ارتباط المشرعين والمسبارين على الموقع الهدف المراد تضخيمه في عينة الدنا DNA أو RNA.
3. يبدأ إنزيم بوليميراز الدنا Taq-Polymerase بالعمل وتضخيم المنطقة بين المشرعين، وحالما يصل الإنزيم إلى منطقة المسبارين، فإنَّ إنزيم البوليميراز له قدرة نزع المسبارين (Exonuclease activity) بالاتجاه 5' إلى 3'، مما يؤدي إلى فصل المسبارين عن بعضهما، ويزيد من الفلورة التي يجري قياسها ليزرياً.
4. يقيس جهاز الـ RT-PCR الفلورة وازدياد شدتها مع كل دورة تضخيمية بحسب العلاقة الأسية لتضخيم الدنا، وذلك أثناء الطور الأسيّ للتضخيم Exponential phase. ثم يجري تعيين التركيز النسبيّ للدنا عبر إسقاط قيم الفلورة على عدد دورات التضخيم بحسب المقياس اللوغاريتمي.
5. يقيس جهاز الـ RT-PCR كمية الـ RNA
أو الـ DNA عبر مقارنة النتائج مع خطٍ معياريٍّ ينفذه الجهاز عبر استخدام سلسلةٍ ممددةٍ معيارية معروفة التركيز من الـ RNA أو الـ DNA.
تُستخدمُ هذه الطريقة في مجاليّ البحث العلميّ والتشخيص الجزيئيّ إذ يشمل استخدامها:
1. التشخيصٌ الكميُّ والسريعٌ للأحماض النووية ذات الصلة بالأمراض الجرثومية المعدية وأمراض السرطان والاضطرابات الوراثية.
2. تحري وكشف أمراضٍ جديدة مثل أمراض الأنفلونزا الخطيرة والجديدة (الطيور والخنازير).
3. التقدير الكمي للتعبير عن الجين Gene expression، حيث بالإمكان معرفة مدى التبدل في التعبير الجيني مع عامل الزمن. وخاصةً لدى فحص وتطبيق أدوية جديدة على مزارع نسيجية ومدى تمايز هذه الخلايا مع وجود عامل مؤثرٍ جديد.
|
4- تعيين تعدد الأشكال النوكليوتيدية المفردة
Single Nucleotide Polymorphisms (SNPs)
يحتوي المجين البشري تقريباً على 3x109 شفع من النوكليوتيدات في الـ DNA، تشكل 23 شفعاً من الصبغيات 22 شفعاً صبغياً جسدياً Autosomes وشفعاً واحداً من الصبغيات الجنسية X وY. لقد جرى الاقتراح منذ أمدٍ طويلٍ بأن معرفة تتالي النوكليوتيدات في المجين البشري سوف يساعد على فهم وتحري الأمراض الوراثية البشرية. لقد ظهرت طرق تحليل تتالي نوكليوتيدات الدنا في منتصف وأواخر سبعينيات القرن المنصرم، حيث بدأت عمليات نشر أبحاث عن تحليل تتالي النوكليوتيدات البشرية لمورثات هنا وهناك. ولقد اُقترح خلال الثمانينات البدء بتحليل تتالي النوكليوتيدات في المجين البشري، والذي بدأ فعلاً بعد عقدٍ من الزمان 1990. لقد ساهم اكتشاف آليات التحليل الآلي المؤتمتة الخاصة بتحليل المجين البشري في الكشف عن تتالي أكثر من ثلاثة ملياراتٍ من النوكليوتيدات المكونة للدنا البشريّ. ولاحقاً تكشفت الخرائط الوراثية المفصلة لتوضع المورثات البشرية في المجين البشري، حيث جرى إنجاز الخَريطَة الوراثية البشرية. وبتعاضد العديد من المؤسسات البحثية العلمية في أنحاء المعمورة، جرى نشر تتالي المجين البشري الأوليّ في عام 2001. إن العدد الفعليّ للجينات المتضمنة في المجين البشري غير محدد بعد. وأشارت التقديرات الأولية لعدد الجينات في المجين البشري من 70,000 إلى 100,000 جين. على كل حال، وفي الوقت الراهن يقدر عدد الجينات البشرية بين 30,000 إلى 40,000 جين. إن التحليل الأولي للمجين البشري أظهر وجود إختلافات مهمةٍ بين الأفراد، ومنها زيادة بمعدل 1,1-1,4 مليون تعدد أشكال نوكليوتيدٍ مفردٍ (SNPs) single nucleotide polymorphisms متوزعة في ثنايا المجين البشريّ. ولايزال العمل جارٍ في تمحيص ودراسة المجين البشري بغرض تعيين هوية وخصائص الجينات البشرية وبخاصة الـ SNPs وتعدد أشكال جينية أخرى ضمن المجين البشري (http//www.nhgri.nih.gov/). لقد أُعلن عام 2003 أنَّ المجين البشريَّ قد أنجز بشكلٍ كامل فيما يخص تتالي نوكليوتيدات الجينات، وأنه يجري الإعداد لدراساتٍ جديدةٍ تخص المجين البشري.
إن أشمل وأضمن طريقة لتعيين تعدد الأشكال النوكليوتيدية المفردة هي تعيين التتالي النوكليوتيدي المباشر لعدد محدود من الأفراد، في حين أن تطبيقها على أعداد كبيرة أو على جينات ضخمة يعد أمراً مكلفاً ومجهداً في الوقت نفسه، لذلك كان لابد من البحث عن طرق أكثر قابلية للتطبيق وتوفر في الوقت والكلفة اللازمة.
تعدد الأشكال النوكليوتيدية المفردة والأمراض SNPs and Diseases
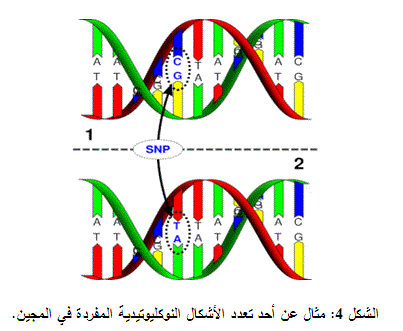
يعد تعيين اختلافات المادة الوراثية DNA variation وكذلك الطفرات الجينية Gene mutations التي تساهم أو تحدد المرض من الأبحاث التي يتم التركيز عليها في الوقت الحالي. إن معظم تعدد الأشكال الجينية على مستوى الـ DNA هي تبدلات أو تعدد أشكال نوكليوتيدية مفردة (SNPs) (Chanock, 2001 وBotstein and Risch, 2003)، منها على سبيل المثال لا الحصر: التنوع الواسع لمستضدات الكريات البيض البشرية (HLA) human leucocyte antigens، النظائر المختلفة (الألليلات) لنظام المستضد الدموي، التنوعات الوراثية المختلفة المترافقة باضطرابات التخثر والمرض القلبي الوعائي مثل العامل الخامسG1691A (Leiden)، والعامل الثاني G20210A، أو طفرات methylene tetrahydrofolate reductase C677T mutations (الشكل 4).
كما يستخدم تعدد الأشكال النوكليوتيدية المفردة في: رسم الخرائط الوراثية للطفرات المسببة للأمراض ( الشكل 5)، اكتشاف الطفرات المسببة للأمراض النادرة وذلك باستخدام رسم الخرائط الوراثية وتعيين مواقع الجينات، بما فيها الطفرات المسؤولة عن أشكال نادرة من انخفاض وارتفاع الضغط، على الرغم من أنه لا زال هنالك تساؤل قيد التحقق في ما إذا كانت الطفرات المسببة لأمراض نادرة قد تشارك أيضاً في أمراض شائعة كارتفاع الضغط الأولي ، ولا تستعمل هذه الطفرات الموصوفة في التحري الروتيني للاختبارات التشخيصية. وكان اكتشاف الطفرات المسببة للتليف الكيسي اكتشافاً لأثر الطفرات في الجينات المسببة على خصائص المرض وتقدمه.
على الرغم من أن تعدد الأشكال النكليوتيدية المفردة المستخدم كأدوات تشخيص مباشرة لا يزال محدوداً، إلا أن هذه الأمثلة الأولية قد تظهر أن الحاجة لهذه الأدوات التشخيصية ستزداد تدريجياً في المستقبل. وفي ما يخص احتياجات هذه الطريقة يمكن لمخابر التشخيص أن تستخدم أعداداً محددةً من تعيينات تعدد الأشكال النكليوتيدية المفردة المجهزة مسبقاً بشكل جيد، وهي عالية الموثوقية ويمكن إجراؤها دون اختصاص واضح في بيئة مخبرية روتينية.
|
|
طرق تنميط تعددات الأشكال النوكليوتيدية المفردة Methods for SNPs typing
الأساليب التقليدية لتنميط الـ SNPs
Traditional approaches of SNPs typing:
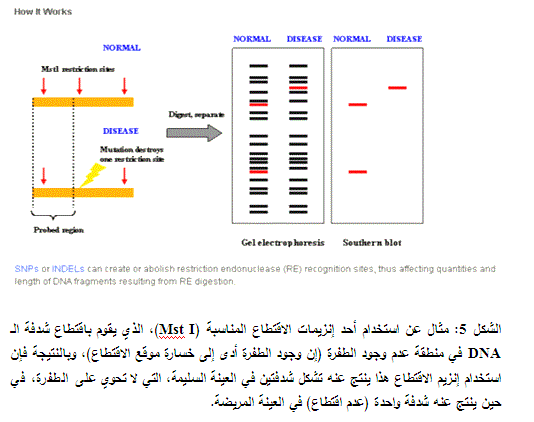
لقد استخدمت عدة أساليب في تنميط الـ SNPs تاريخياً وذلك في مجال البحث العلميّ وفي المخابر السريرية. وإن واحداً من أولى هذه الأساليب في تنميط الـ SNPs هو تقانة مشتركة بين الـ PCRوالـ RLFP ويُشارُ إليها بتقانة تفاعل سلسلة بوليميراز الدنا- تعدد أشكال طول شدف الاقْتِطاع (PCR-RFLP)، والتي يمكن استخدامها في عملية التنميط عندما يخلق الـ SNP أو يلغي موقع قصٍ (قطعٍ) لإنزيم اقْتِطاع الدنا Restriction Enzyme. يجري عادة تطبيق الطريقة عبر تضخيم منطقة مهمة محددة من الدنا بوساطة مَشْرَع تقييد لتلك المنطقة. ثم يلي ذلك تقطيع الدنا الناتج من تفاعل الـ PCR بإنزيم اقْتِطاعٍ مناسبٍ ومن ثم تقصي نواتج ذلك بوساطة الرحلان الكهربائيّ (الشكل 5).
لقد جرى استخدام هذا الأسلوب بشكلٍ شائعٍ، حتى في حالات كون الـ SNP لم يبدل نمط القطع بإنزيم الاقْتِطاع، وذلك عبر استخدام الـ PCR- المتواسط بتطفير الموقع الموجه PCR-mediated site-directed mutagenesis. لقد استخدمت هذه التقانة التقليدية المشتقة من PCR-RFLP مشرعاً معدلاً، بالتساوي مع الـSNP، وذلك لخلق أولإلغاء موقع الاقتطاع الإنزيمي. إنَّ الضعف الأوليّ في هذا الأسلوب يكمن في كثافة العمل المبذول أثناء تنفيذه، بالإضافة إلى حقيقة أنه لايوجد هضم نهائياً أو يوجد هضم غير كاملٍ لنواتج تفاعل الـ PCR، مما قد يؤدي إلى نتائج خاطئة.
على كل حال، إن وجود مواقع قصٍ أو إضافةٍ ذاتية المنشأ أو هندسة/ خلق مواقع قصٍ إضافية ضمن نواتج الـ PCR يمكن أن تساعد كضوابط داخلية في عملية الهضم الإنزيمي بإنزيمات اقتطاع الدنا.
إن تقانة الأليل النوعي للـ PCR Allele-Specific PCR (ASPCR)، والمعروفة بـنظام الطفرة الحرون المضخم Amplification Refractory Mutation System (ARMS) أو الـ PCR مع مشارع التتالي النوعية PCR-sequence-specific primers (PCR-SSP)، تقوم على حقيقة أن إنزيم بلمرة الدنا Taq Polymerase سوف يكون أقل فعالية في بناء مَشرَع قليل الدنا، خاصة عندما تكون النهاية 3' لقطعة المَشرَع غير متطابقة تماماً مع موقعها الهدف في الدنا المُراد تضخيمه. ونتيجةً لذلك، فإنه يمكن تصميم المشارع التي تستبعد الأساس النوكليوتيدي المُستبدل، في حالة الخبن deletion الضئيل، أوالإدخال Insertion. يجري ترتيب التحاليل في مجموعتين تفاعليتين لأجل الأنظمة ثنائية الأليل حيث أن كل تفاعلٍ يستخدم مشارع نوعيةٍ لكشف أليلٍ مفرد. إنَّ نقطة نهاية التحليل هي عادة تحليل نواتج التفاعل بالرحلان الكهربائيّ، وكشف الأحجام المناسبة للنواتج التي تعد دالاّت على وجود الأليلات. إن نواتج تميم التضخيم co-amplification الثاني تستخدم عادةً كضابطٍ داخلي، ولهذا فإن ذلك الفشل التقنيّ هو غير مربكٍ بتداخله مع غياب الأليل. يمكن تحسين النوعية التي تمتاز بها المشارع الأليلية من خلال إدخال مشارع إضافية غير متطابقة في المواقع -2، -3 أوقبل النوكليوتيد الأخير. يؤدي هذا الإجراء الإضافي في عدم استقرار النهاية 3' للمشرع إلى إنقاص عدم انطباق المشرع على دنا المجين المراد تضخيمه وذلك أثناء الدورات الأولى من تفاعل الـ PCR. لقد جاءت أولى التقارير العلمية لتطبيق هذه الطريقة في الكشف عن تبدل مواقع الأسس أدنين إلى ثيمين في جين β-غلوبين البشرية المسؤولة عن فقر الدم المنجلي. وبعد ذلك، استخدم هذا الأسلوب في االتحري بشكلٍ واسعٍ في مجال البحث العلمي والمخابر السريرية، في تنميط زمر المستضدات الدموية والطفرات المترافقة باختطار التجلط/ الخُثار أو أمراض القلب وتنميط الـ HLA، وα1- ضِدُّ الترِيبْسين α1- Antitrypsin، وستيرويد 21- هيدروكسيلاز. إن الجدوى من هذه الطريقة أنها لا تتطلب جهداً مخبرياً كبيراً، كما هو حال طريقة PCR-RFLP ولا تتطلب اقتطاعاً إنزيمياً بعد إجراء تفاعل الـ PCR. على كل حال هنالك حاجة لتوخي الوصول بهذه الطريقة نحو الأمثل. لقد نُشرت تقارير عن حدوث تطورات في هذه الطريقة، ومنها تقانة الـ MLPA. كما جرى تطوير طريقة فائقة تقوم على أن المشارع النوعية لأليل ما قد جرى تذييلها بتتالي مشرعي وذلك لإظهار مزيد من الوضوح في تعدد الأشكال الموجود في المنتج الأولي للـ PCR. وتعد هذه الطريقة استراتيجية نافعة في التنميط الدقيق للمواقع الوراثية المعقدة مثل HLA.
حالياً، في مجال البحث العلمي والتطبيقات المخبرية السريرية، لا توجد طريقة مميزة ومحددة في تنميط الـ SNP. ومن الشائعٌ استخدام تقانات تشمل التهجين وإطالة المَشرَع وطرق الاقتطاع الإنزيمي المترافقة بنظم كشف مختلفة. ولقد طورت عدة شركات تقانات حيوية بأنظمة دقيقة للغاية للتمييز الأليليّ ونظم التفاعلات وطرق التحري والكشف. |
| الرقائق المِكْرَوِيّة Affymetrix Microchips: |
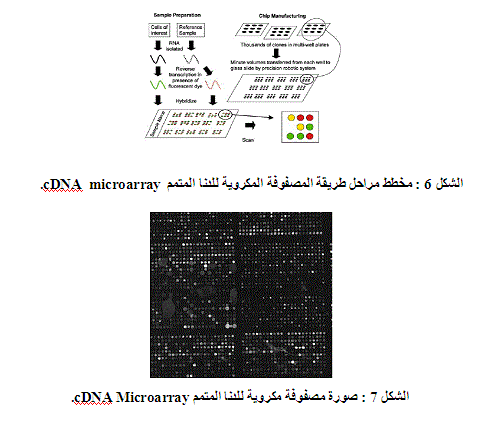
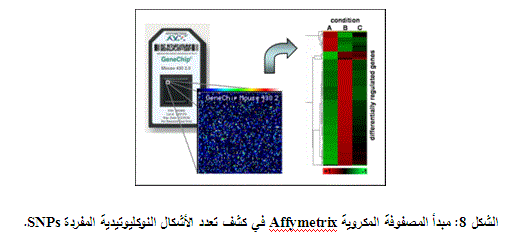
هي طريقة مميزة طورتها شركة Affymetrix، تقوم على مبدأ تهجين الحمض النووي على سطح صلب يحوي أشرطة قصيرة من الدنا DNA كمتعددات نوكليوتيدية، بحيث تمكن الطريقة من إجراء التنميط الوراثي المتزامن لعدد كبير من تعدد الأشكال النوكليوتيدية المفردة، بالإضافةً إلى إمكانية استخدامها في تحليل التعبير الجيني. تعتمد التقانة على دمغ شبكة من النوكليوتيدات القصيرة المصنعة على شريحة مجهرية زجاجية باستخدام طريقة الحفر على الزجاج بالليزر، بحيث تخدم هذه الرقاقة كقاعدة لتستقبل النوكليوتيدات المضافة في المراحل التالية (الشكل 6) وذلك بعد تحفيز المواقع المختارة على الرقاقة بشكل صحيح وهي المرحلة التي تمثل الخطوة الحرجة في هذه الطريقة. ولتحديد النمط الجيني يجري تصنيع نوكليوتيدات قصيرة متتامة مع تعدد الأشكال النوكليوتيدية المفردة قيد الاهتمام وذلك باستخدام النوكليوتيدات الأربعة جميعها.
تكمن قوة هذه الطريقة في التحليل المتواقت لعدد كبير من تعدد الأشكال النوكليوتيدية المفردة، ويمكن استخدامها، في الدراسات البحثية، لتحليل عدد ثابت من تعدد الأشكال في كامل المجين البشري (الشكلان 7 و8).
|
|
| وفي مجال التشخيص، لقد جرى تصميم رقاقات مِكْرَوِيّة لها القدرة على تحليل كم كبير من تعدد الأشكال النوكيوتيدية المفردة SNPs كجزء من التحليل الشامل لتعددات الأشكال جميعها. |
مثيلة الدنا DNA Methylation
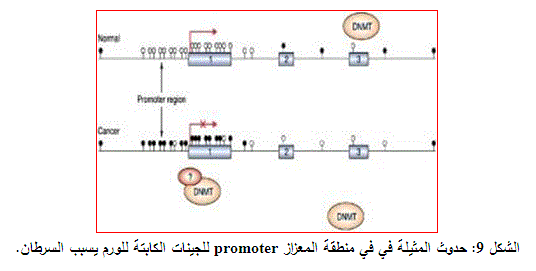
تعد مثيلة الدنا DNA من العمليات الحيوية التي تحصل في جسم الإنسان بشكل طبيعي، حيث يجري خلالها إضافة جذر الميثيل إلى ذرة الكربون الخامسة في السيتوزين (واحد من النوكليوتيدات المكونة للـ DNA) وتحدث في المناطق الغنية بـ CpG (السيتوزين – الفسفور- الغوانين) ويؤدي ذلك إلى تخميد الجين وتعطيلها عن العمل. أما إذا حدثت هذه التعديلات في منطقة المِعْزاز promoter للجينات المسؤولة عن تصحيح الأخطاء الحاصلة خلال مراحل نسخ وتضاعف DNA، والجينات المسؤولة عن مرحلة الاستماتة الخلوية apoptosis والتي يطلق عليها اسم الجينات الكابتة للورم Tumor suppressor genes فإن ذلك يؤدي إلى تشكل الخلايا السرطانية (الشكل 9).
|
 |
لقد مرت فترة من الزمن جرى أثناءها استبعاد فكرة مثيلة الدنا كحدث مسبب للسرطان، ويعود ذلك إلى صعوبات مرتبطة بتحليل الدنا المُمَثيل. جرى حديثاً تطوير العديد من الطرق التي تعتمد على PCR للكشف عن المثيلة في منطقة المعزز للجينات الكابتة للورم وبالتالي إمكانية الكشف عن وجود خلايا سرطانية ونذكر منها:
1. Methylation Specific PCR (MSP).
2. Methylation-Sensitive (Restriction Enzyme PCR).
3. Real-Time PCR methodology.
في الطريقة الأولى تجري إضافـة Bisulfite إلى DNA والتي تعمل على تحويل السيتوزين الخالي من جذر الميثيل إلى اليوراسيل، بعدها يضاف المشرع Primer الخاص بالمنطقة المراد تحليلها إلى DNA المعالج بالـ Bisulfite ليحصل تلدين annealing بين المشرع وDNA، فقط في حالة وجود جذر الميثيل وسوف يظهر شريط على هلامة الآغاروز.
تعتمد الطريقة الثانية على إضافة إنزيمات (تميز بين DNA المُمَثيل وغير الممثيل) إلى DNA، تعمل الإنزيمات على اقتطاع DNA، باستثناء المناطق التي تحوي على جذر الميثيل. يضاف بعدها مشرع Primer خاص بالمنطقة المراد تحليلها إلى DNA فيحدث تلدين annealing بين المشرع والـ DNA الحاوي على جذر الميثيل وسوف يظهر لدينا شريط على هلامة الآغاروز يدل على وجود المثيلة في DNA (الشكل 10). تمتاز الطريقتان الأولى والثانية بإمكان الكشف عن المثيلة نوعياً.
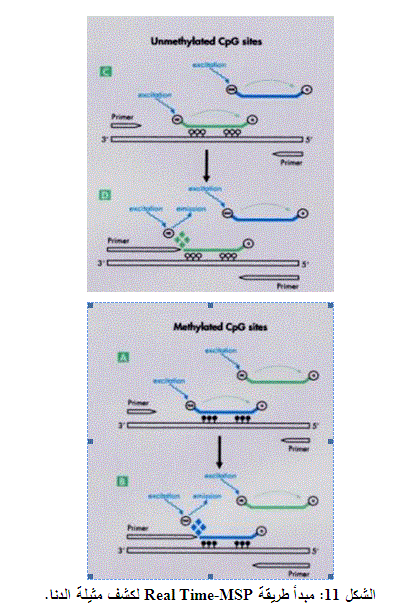
تستخدم الطريقة الثالثة مبدأ Real Time- MSP (Methylation-Sensitive Polymerisation) حيث تكون الخطوة الأولى معالجة DNA بـ Bisulfite والذي يعمل على تحويل السيتوزين الخالي من جذر المثيل إلى اليوراسيل، ومن ثم إضافة مسبارين أحدهما خاص بالمثيلة، يرتبط بـ DNA فقط في حالة وجود جذر الميثيل، بينما يرتبط المسبار الثاني بـ DNA الخالي من جذور الميثيل (الشكل11). وبما أن كلا المسبارين يحمل قسماً مفلوراً، يقوم الجهاز Real Time PCR بإعطاء كمية DNA الحاوي على جذر الميثيل وكمية DNA الخالي من جذور الميثيل، لذا تمتاز هذه الطريقة بإمكانية الكشف عن المثيلة نوعياً وكمياً، الأمر الذي يساعد الطبيب على متابعة مدى تجاوب المريض مع المعالجة وتحديد نجاحها. تعد هذه التقنية من التقنيات الواعدة في الكشف المبكر عن الخلايا السرطانية.
أظهر البحث العلمي مؤخراً أن MeCP2 هو أحد أفراد عائلة بروتينية تميز بشكل انتقائي تجمعات CpG الممثيلة وإن ارتباط هذه البروتينات بالدنا يؤدي إلى تعديل بنية الكروماتين، مما يؤدي إلى استبعاد تعبير الجين عن ذاتها، وقد طورت شركة Panomics طريقة جديدة لتحليل وتحري حالة مثيلة الدنا وتقوم الطريقة على مبدأ ارتباط MeCP2، الذي يعمل على التمييز بين المجموعات الممثيلة وغير الممثيلة للمعززات الجينية. وهذه الطريقة سهلة جداً ومباشرة إذا ما قورنت مع الطريقة المعتمدة على الصوديوم ثنائي السولفيت للنوكليوتيدات الممثيلة (الشكل 12).
|

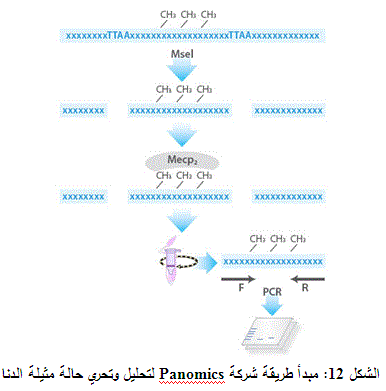
|
| مقايسة ربط قليلات النكليوتيدات (OLA)
Oligonucleotide Ligation Assay
|
طريقة ملبا (MALPA)
Multiplex Ligation- dependent Probe Amplification
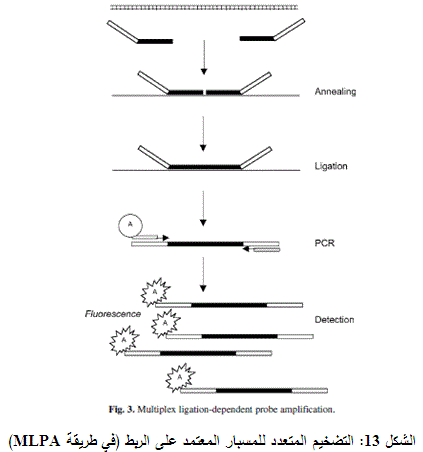
هي طريقة تعتمد على استنساخ عدة مواقع في الشريط الوراثي باستخدام تفاعل الـ PCR وهذا ما يدعى بالـ Multiplex PCR من خلال التهجين بين الشريط الوراثي المدروس وبين المسابير Probes المصممة لجين معين. يتألف كل مسبار من قطعتين تلتحمان لاحقا أثناء التهجين ويعد المسبار الملتحم كقالب أثناء تفاعل الـ PCR. يستخدم في هذا التفاعل فقط مشرعان Primers متخصصان بنهايات المسابير المستخدمة جميعها، ويكون أحدهما موسوماً labeled. إن عملية وسم المشرع ستساعد في تقدير كمي لعدد نسخ الـ DNA البدئية وعدد النسخ المنتجة بعد تفاعل الـ PCR. تصمم هذه المسابير بأطوال مختلفة بحيث يسهل تمييزها بعد الرحلان.
هذه الطريقة حساسة لأي تبدل (حذف أو إضافة) وخاصة عند اختبار مناطق التغيير في منطقة الجين أو المناطق المرمزة ضمن الجين والتي تدعى بالإكسونات Exons. تستخدم هذه التقنية من أجل التعرف على حالة مثيلة في منطقة الجين، تحديد تعدد أشكال الأسس النوكليوتيدية المفردة SNPs.
يتلخص التفاعل في طريقة ملبا كما يلي:
• يتطلب كل تفاعل لأكسون معين وجود مسبارين.
• في مرحلة التهجين تُلدن (تلتصق) هذه المسابير بالمنطقة المتخصصة على الأكسون الهدف Annealing.
• يلتصق المسباران بعملية الربط ligation.
• تستخدم هذه المسابير كقالب في تفاعل الـ PCR (الشكل 13).
تختلف هذه المسابير في الطول وتكون عادة بين 130 و490 أساس آزوتي، ويتم تحديد النتائج بالمقارنة النسبية بين ارتفاعات الذرى الناتجة لكل من الشخص المريض والشخص السليم. فعلى سبيل المثال، إن 65% من الخلل المتسبب في المورثة DMD المسؤولة عن حثل دوشن/ بيكر العضلي ناجم عن طفرات خبن أو تضاعف في منطقة الإكسونات.
مميزات طريقة MLPA أنها طريقة سريعة وغير مكلفة نسبياً وبسيطة. الميزة الرئيسة في هذه الطريقة هي استهداف ما يقارب خمسين منطقة أكسون في تفاعل واحد، كما أن احتياجاتها من المادة الوراثية قليلة جداً مقارنة بالطرق الأخرى، يمكن استخدام المسابير المصممة للعديد من التطبيقات المتاحة تجارياً، ويحتاج تطبيق هذه الطريقة إلى جهاز PCR وجهاز Sequencer. ولقد أصبحت هذه الأجهزة متوفرة في معظم المخابر المتخصصة في التحاليل الوراثية، وبالتالي فهي لا تحتاج إلى جهاز خاص بها، كما يمكن الحصول على المعلومات خلال 24 ساعة ولــ 96 عينة. أحد تطبيقات ملبا هو إمكان كشف مثيلة الدنا، وتختلف هذه الطريقة عن طريقة ملبا العادية التي تحتوي على مرحلة هضم بإنزيم متخصص حساس للميثيل.
عيب هذه الطريقة هو أن هنالك شركة واحدة تنتجها وهي تحتاج دوماً إلى وجود مكررات وعينات مرافقة كشاهد، كما أنها طريقة حساسة لكمية ونوعية المادة الوراثية، لذلك يفضل استخدام أكثر من عينة للمريض أثناء الفحص. كما أن وجود خبن لأساس نوكليوتيدي واحد في منطقة التحام المسبار قد ينتج عنه فشل عملية التهجين وتظهر على شكل خبن كلي للمنطقة المستهدفة. لذلك ولتأكيد منطقة الخبن، هنالك حاجة لاستخدام تقنيات أخرى يجب أن تطبق لدعم نتائج تقنية MLPA مثل real- time PCR,، Southern blot...إلخ، أو حتى فحص الترميز الوراثي للمنطقة كاملة. |
 |
Microsatellites السواتل المِكْرَوِيّة
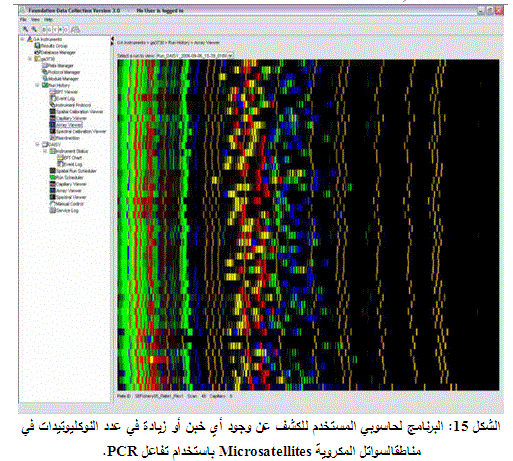
السواتل المِكْرَوِيّة هي مناطق من DNA تحوي على تكرار من النوكليوتيدات وهذه التكرارات تختلف من شخص إلى آخر. لدى الإنسان يكثر التكرار التالي (CA)n والذي يحدث آلاف المرات عبر المجين البشري، ويعد طول هذه المناطق كعلامة مميزة يمكن استخدامها في التحليل الطبي للكشف عن الكثير من الأمراض وخاصة السرطان، فعادةً ما تترافق الخلايا السرطانية بتبدل كبير في عدد التكرارات عند مقارنتها مع الخلايا الطبيعية، ويعود السبب في ذلك إلى خلل في عمل جينات الإصلاح مؤدياً إلى زيادة أو نقص في عدد التكرارات وهذه التكرارات لها دور رئيسي في عملية إصلاح الخطأ الحاصل أثناء عملية نسخ وتضاعف DNA بالإضافة إلى دورها في الاستماتة الخلوية، وأي خلل في عمل هذه الجينات يؤدي إلى ضعف قدرة الخلية على إصلاح DNA، مما يؤدي إلى تراكم الطفرات وتشكل الورم ونشوء ظاهرة تُدعى عدم ثبات التكرارات النوكليوتيدية (MSI: Microsatellite Instability) (الشكل 14). أشارت الدراسات التي أجريت مؤخرًا إلى أن اختبار MSI يعد استراتيجية فعالة لتحديد الأفراد الواقعين تحت اختطار الإصابة بسرطانة القولون والمستقيم الوراثي عديمة السلائل (HNPCC) Hereditary Nonpolyposis Colorectal Carcinoma وكواسم ممتاز وسهل لتقيم وجود خلل في نظام جينات الإصلاح. حيث يقوم الأخصائيون بإجراء اختبار MSI على القولون كخطوة أولى في الاختبار الجيني، للعائلات ذات التاريخ الطبي الموحي بوجود HNPCC، لأن حوالي 90% من أمراض سرطان القولون متعلقة بمتـلازمة HNPCC والتي لها تذبذب عالٍ في مناطق السواتل المِكْرَوِيّة microsatellites.
يتألف اختبار MSI من خمسة واسمات تستخدم في الكشف عن وجود أي خبن أو زيادة في عدد النوكليوتيدات في مناطق microsatellites وذلك باستخدام تفاعل PCR، ويتم تحليل النواتج بوساطة الرحلان الكهربائي الشعري باستخدام جهاز ABI 310 Genetic analyzer حيث يعد الورم ذا عدم ثباتية عالية إذا وجدت تبدلات في اثنان أو أكثر من المناطق، وذا عدم ثباتية منخفضة إذا وجدت تبدلات في منطقة واحدة، وثابتاً إذا لم تكن هنالك تبدلات بين خلايا المريض الورمية والخلايا الطبيعية.
أخيراً لقد ساهمت طريقة الـ PCR والطرق الجزيئية الأخرى في إحداث ثورة في تحليل المجين البشري. وقد كان ذلك مرئياً بشكل واضح في التطور السريع في الطرق التي تستخدم مبدأ PCR في تحري وكشف الطفرات المسببة للأمراض الوراثية البشرية. لقد ساعد التقدم المذهل في هذا المجال الأطباء على التأكد من تشخيص حالات صعبة التشخيص سريرياً، لاسيما التشخيص قبل الولادة وتحليل استعداد الفرد للإصابة بمرض وراثي معين. كما وتعد طرق كشف الطفرات المسببة لمقاومة الفيروسات للأدوية بمثابة حجر الزاوية في علاج الأمراض الفيروسية مثل HCV و HIV. وإنه من المتوقع، مع المزيد من التطوير في طرق التحليل الآلي الموصوفة في هذه المراجعة، التوصل إلى المزيد من التسهيلات لا سيما في مجال الاستخدام المعتاد لاختبارات التشخيص السريري والتطبيقات السريرية في الوراثة الجزيئية والسرطان (الشكل 15).
|

|
| المراجع References |
1.Hall N (May 2007). "Advanced sequencing technologies and their wider impact in microbiology". J. Exp. Biol. 210 (Pt 9): 1518–25.
2.Church GM (January 2006). "Genomes for all". Sci. Am. 294 (1): 46–54.
3.Schuster, Stephan C. (2008). Next-generation sequencing transforms today's biology. 5. Nature Methods. pp. 16–18
4. Aarskog, N. K. and Vedeler, C. A. Real-time quantitative polymerase chain reaction.
Hum. Genet. 107:494–498, 2000.
5. Yang, J. H., Lai, J. P., Douglas, S. D., Metzger, D., Zhu, X. H., and Ho, W. Z. Real-time RT-PCR for quantitation of hepatitis C virus RNA. J. Virol. Methods 102:119–128, 2002.
6. Emig, M., Saussele, S., Wittor, H., et al. Accurate and rapid analysis of residual disease in patients with CML using specific fluorescent hybridization probes for real time quantitative RT-PCR. Leukemia 13:1825–1832, 1999.
7. Wittwer, C. T., Herrmann, M. G., Gundry, C. N., and Elenitoba-Johnson, K. S. Real-time multiplex PCR assays.
Methods 25:430–442, 2001.
8. van der Velden, V. H., Hochhaus, A., Cazzaniga, G., et al Detection of minimal residual disease in hematologic malignancies by real-time quantitative PCR: principles, approaches, and laboratory aspects. Leukemia 17:1013–1034, 2003.
9. Donohoe, G. G., Laaksonen, M., Pulkki, K., Ronnemaa, T., and Kairisto, V. Rapid single-tube screening of the C282Y hemochromatosis mutation by real-time multiplex allele-specific PCR without fluorescent probes. Clin. Chem. 46:1540–1547, 2000.
10. Neoh, S. H., Brisco, M. J., Firgaira, F. A., Trainor, K. J., Turner, D. R., and Morley, A. A. Rapid detection of the factor V Leiden (1691 G > A) and haemochromatosis (845 G > A) mutation by fluorescence resonance energy transfer (FRET) and real time PCR.
J. Clin. Pathol. 52:766–769, 1999.
11. Wabuyele, M. B., Farquar, H., Stryjewski, W., et al. Approaching real-time molecular diagnostics: single-pair fluorescence resonance energy transfer (spFRET) detection for the analysis of low abundant point mutations in K-ras oncogenes. J. Am. Chem. Soc. 125:6937– 6945, 2003.
12.Redeker, E.J., et al. (2008). Multiplex ligation-dependent probe amplification (MLPA) enhances the molecular diagnosis of aniridia and related disorders Mol Vis 14, 836-840.
13.Jeuken, J., et al. (2007). MS-MLPA: an attractive alternative laboratory assay for robust, reliable, and semiquantative detection of MGMT promoter hypermethylation in gliomas. Lab Invest. 87(10):1055-65.
14.Jeuken, J., et al. (2006).
Multiplex ligation-dependent probe amplification: a diagnostic tool for simultaneous identification of different genetic markers in glial tumors J Mol Diagn 4, 433-443.
15. Delahunty, C. M., Ankener, W., Brainerd, S., Nickerson, D. A., and Mononen, I. T. Finnish-type aspartylglucosaminuria detected by oligonucleotide ligation assay. Clin. Chem. 41:59–61, 1995.
16. Brinson, E. C., Adriano, T., Bloch, W., et al. Introduction to PCR/OLA/SCS, a multiplex DNA test, and its application to cystic fibrosis. Genet. Test. 1:61–68, 1997.
17. Grossman, P D; Bloch W; Brinson E. et al.
High-density multiplex detection of nucleic acid sequences: oligonucleotide ligation assay and sequence-coded separation.
Nucleic Acids Res. 22:4527–4534, 1994.
18. Schouten, J. P., McElgunn, C. J., Waaijer, R., Zwijnenburg, D., Diepvens, F., and Pals, G. Relative quantification of 40 nucleic acid sequences by multiplex ligation-dependent probe amplification. Nucleic Acids Res. 30:e57, 2002.
19.Loukola A., et al., 1999. Microsatellite Instability in Adenomas as a Marker for Hereditary Nonpolyposis Colorectal Cancer. American Journal of Pathology. 1999;155:1849-1853.
20.Jass JR, Stewart SM, Stewart J, Lane MR: Hereditary nonpolyposis colorectal cancer: morphologies, genes and mutations. Mutat Res 1994, 310:125-133.
21.Aaltonen LA, et al., de la Chapelle A: Replication errors in benign and malignant tumors from hereditary nonpolyposis colorectal cancer patients. Cancer Res 1994, 54:1645-1648
22.http://www.ncbi.nlm.nih.gov/projects/genome/probe/doc/TechRFLP.shtml
23.http://www.affymetrix
24.http://www.mrcholland.com/WebForms/WebFormMain.aspx?Tag=zjCZBtdOUyAt3KF3
25. Whitcombe, D., Kelly, S., Mann, J., Theaker, J., Jones, C., and Little, S. Scorpion primers—a novel method for use in single tube genotyping. Am. J. Hum. Genet. 65:2333, 1999.
26. Wu, D. Y., Ugozzoli, L., Pal, B. K., and Wallace, R. B. Allele-specific enzymatic amplification of beta-globin genomic DNA for diagnosis of sickle cell anemia. Proc. Natl. Acad. Sci. USA 86:2757–2760, 1989.
27. Sommer, S. S., Cassady, J. D., Sobell, J. L., and Bottema, C. D. A novel method for detecting point mutations or polymorphisms and its application to population screening for carriers of phenylketonuria. Mayo Clin. Proc. 64:1361–1372, 1989.
28. Hogervorst, F. B., Nederlof, P. M., Gille, J. J., et al. Large genomic deletions and duplications in the BRCA1 gene identified by a novel quantitative method. Cancer Res. 63:1449–1453, 2003.
29. Nakagawa, H., Hampel, H., and de la Chapelle, A. Identification and characterization of genomic rearrangements of MSH2 and MLH1 in Lynch syndrome (HNPCC) by novel techniques. Hum. Mutat. 22:258, 2003.
30. Cutting, G. R., Kasch, L. M., Rosenstein, B. J., et al. A cluster of cystic fibrosis mutations in the first nucleotide-binding fold of the cystic fibrosis conductance regulator protein.
Nature 346:366–369, 1990.
31. Vohl, M. C., Couture, P., Moorjani, S., et al. Rapid restriction fragment analysis for screening four point mutations of the low-density lipoprotein receptor gene in French Canadians. Hum. Mutat. 6:243–246, 1995.
32. Sommer, S. S., Groszbach, A. R. and Bottema, C. D. PCR amplification of specific alleles (PASA) is a general method for rapidly detecting known single-base changes. Biotechniques 12:82–87, 1992.
33. Hessner, M. J., Luhm, R. A., Pearson, S. L., Endean, D. J., Friedman, K. D., and Montgomery, R. R. Prevalence of prothrombin G20210A, factor V G1691A (Leiden), and methylenetetrahydrofolate reductase (MTHFR) C677T in seven different populations determined by multiplex allele-specific PCR. Thromb. Haemost. 81:733–738, 1999.
34. Hessner, M. J., Budish, M. A., and Friedman, K. D. Genotyping of factor V G1691A (Leiden) without the use of PCR by invasive cleavage of oligonucleotide probes. Clin. Chem. 46:1051–1056, 2000.
35. Raife, T. J., Lentz, S. R., Atkinson, B. S.,Vesely, S. K., and Hessner, M. J. Factor V Leiden: a genetic risk factor for thrombotic microangiopathy in patients with normal von Willebrand factor-cleaving protease activity.
Blood 99:437–442, 2002.
36. Dinauer, D. M., Luhm, R. A., Uzgiris, A. J., Eckels, D. D., and Hessner, M. J.
Sequence-based typing of HLA class II DQB1. Tissue Antigens 55:364–368, 2000.
37. Mohr, S., Leikauf, G. D., Keith, G., and Rihn, B. H. Microarrays as cancer keys: an array of possibilities.
J.Clin. Oncol. 20:3165–3175, 2002.
38. Bain, B. J. Routine and specialised techniques in the diagnosis of haematological neoplasms. J. Clin. Pathol. 48:501–508, 1995.
39. Kepler, T. B., Crosby, L., and Morgan,
K. T. Normalization and analysis of DNA microarray data by self-consistency and local regression. Genome Biol. 3:RESEARCH0037, 2002.
40. Robertson, K. D. and Wolffe, A. P. DNA methylation in health and disease. Nat. Rev. Genet. 1: 11–19, 2000.
41. Ehrlich, M. DNA hypomethylation, cancer, the immunodeficiency, centromeric region instability, facial anomalies syndrome and chromosomal rearrangements.
J. Nutr. 132:2424S–2429S, 2002.
42. Gibbons, R. J. and Higgs, D. R. Molecular-clinical spectrum of the ATR-X syndrome. Am. J. Med. Genet. 97:204–212, 2002.
43. Gibbons, R. J., McDowell, T. L., Raman, S., et al. Mutations in ATRX, encoding a SWI/SNF-like protein, cause diverse changes in the pattern of DNA methylation.
Nat. Genet. 24:368–371, 2000.
44. Oberle, I., Rousseau, F., Heitz, D., et al. Instability of a 550-base pair DNA segment and abnormal methylation in fragile X syndrome.
Science 252:1097–1102, 1991.
45. Kremer, E. J., Pritchard, M., Lynch, M., et al. Mapping of DNA instability at the fragile X to a trinucleotide repeat sequence p(CCG)n. Science 252:1711–1714, 1991.
46. Baumer, A. Analysis of the methylation status of imprinted genes based on methylation-specific polymerase chain reaction combined with denaturing high-performance liquid chromatography.
Methods 27:139–143, 2002.
47. Couvert, P., Poirier, K., Carrie, A., DHPLC-based method for DNA methylation analysis of differential methylated regions from imprinted genes.
Biotechniques 34:356–362, 2003.
48. Hemminki, A., Mecklin, J. P., Jarvinen, H., Aaltonen, L. A., and Joensuu, H. Microsatellite instability is a favorable prognostic indicator in patients with colorectal cancer receiving chemotherapy.
Gastroenterology 119:921–928, 2000.
|
| |
| المجلد 6 ,
العدد 2
, رجب 1432 - تموز (يوليو) 2011 |
|
|
|
