| الملخص Abstract |
| تعد التعددات الشكلية الجينية وحيدة النوكليوتيد Single Nucleotide Polymorphisms (SNPs) من الواصمات الجينية المستحبة. ولقد جرى تطوير تنوع ضخم من التقانات لتحري هذا النمط من الطفرات. لكن غالباً ما تكون هذه التقانات باهظة التكلفة وتتطلب معدات خاصة.
قمنا في دراستنا بتقييم لطريقة (ARMS-PCR) Amplification Refractory Mutation System-Polymerase Chain Reaction. وأُجريت عملية الأمثلة باستخدام عينات دنا جينومي لمرضى التهاب مفاصل روماتويدي (RA) للكشف عن التعدد الشكلي (G/T) STAT4 rs7574865. اشتملت الدراسة على 13 عينة دنا DNA، التي سبق وأخضِعت إلى تفاعل سلسلة الدنا لمعرفة النمط الجيني لـ STAT4 rs7574865.
جرى تحديد الشروط المثلى لتفاعل ARMS-PCR باستخدام تراكيز مشارع مختلفة ودرجات حرارة تلدين متباينة. وبتطبيق تفاعل ARMS-PCR بشروطه المؤمثلة على العينات المدروسة، أظهرت عينتان منهم نمطاً جينياً مختلفاً عن النمط الظاهر بتفاعل السَلْسَلة.
توصلت دراستنا إلى الشروط الحرارية وتراكيز المشارع لإجراء تفاعل ARMS-PCR، من أجل التنميط الجيني للتعدد الشكلي STAT4 rs7574865، لكنها لا تزال تحتاج لبعض التعديلات للوصول إلى نوعية أكثر. فالـ ARMS-PCR طريقة سهلة قابلة للتطبيق وغير مكلفة.
|
Single Nucleotide Polymorphisms (SNPs) are becoming favored genetic markers. A large variety of the techniques have been developed for screening this type of mutations. But mostly, these techniques are expensive and require specialized equipments.
In our study, we worked on evaluating ARMS-PCR (Amplification Refractory Mutation System-Polymerase Chain Reaction) method. The optimization process was done using genomic DNA samples from rheumatoid arthritis patients to detect the genotype STAT4 rs7574865 (G/T).
The study included 13 DNA samples, whose STAT4 rs7574865 genotypes were already known using DNA sequencing method.
The optimization was done using different primers concentrations and annealing temperatures. The optimized ARMS-PCR reaction was applied to the samples, but two of them showed a different genotype from the genotype shown by sequencing reaction.
Our study has set the thermal conditions and primer concentrations for the ARMS-PCR reaction, for the genotyping STAT4 rs7574865 polymorphism, but it still needs some modifications to attain more specificity. ARMS-PCR is an easy, applicable and not costly method.
|
| المقدمة Introduction |
تعد التعددات الشكلية الجينية وحيدة النوكليوتيد (SNPs) Single Nucleotide Polymorphisms من الواصمات الجينية Genetic markers واسعة الاستخدام في العديد من المجالات، ولاسيما في مجال تحري الألائل المرتبطة مع اختطارالإصابة بمرض ما (1).
جرى تطوير العديد من التقانات والطرق لتحري هذا النوع من الطفرات ولاسيما بعد التقدم الهائل الحاصل في مجال التقانة الحيوية، ولكن غالباً ما تكون هذه الطرق باهظة التكلفة وتحتاج إلى معدات ضخمة، كتقانات سلسلة الدنا DNA sequencing، أو أن تتطلب استعمال إنزيمات Nucleases نوعية بشرط أن تقطع في مكان وجود الطفرة (2). جرى في دراستنا تقييم لطريقة (ARMS-PCR) Amplification Refractory Mutation System-Polymerase Chain Reaction يعتمد مبدأ الطريقة على إجراء تفاعلَي PCR تقليديين Conventional PCR في أنبوبين منفصلين أحدهما باستعمال مشرع متمم للمنطقة المدروسة نوعي للطفرة عند النهاية 3' mutant specific primer، وفي الأنبوب الآخر مشرع متمم للمنطقة المدروسة نوعي للنمط الشائع عند النهاية 3' wild type specific primer، إضافةً إلى مشرع مشترك common primer في كلا الأنبوبين، ثم الكشف عن نواتج التفاعل بإجراء الرحلان الكهربائي على الهلامة Gel electrophoresis، حيث تظهر عصابة دالة على ناتج تفاعل الـ PCR في حال تتام الدنا مع المشرع النوعي في الأنبوب (1).
جرى التقييم باستخدام عينات دنا جينومي مأخوذة من مرضى التهاب مفاصل روماتويدي Rheumatoid Arthritis (RA) للكشف عن التعدد الشكلي (G/T) STAT4 rs7574865 المرتبط بالإصابة بالـ (RA) (3، 4)، حيث أن النمط الطافر من هذا التعدد الشكلي يؤدي إلى زيادة مستويات التعبير عن بروتين STAT4 الضالع في سبيل إمراضية الـ RA (5).
وبالرغم من أن ARMS-PCR كانت قاصرة عن كشف هذا التعدد فقد كانت طريقة سهلة وسريعة ورخيصة مقارنة بغيرها من الطرق.
|
| المواد والطرق |
الاعتيان
جُمعت عينات الدراسة من مرضى التهاب مفاصل روماتويدي مراجعين لعيادة المفاصل الخارجية في مستشفى ابن النفيس بدمشق، أو من المقبولين في شعبة المفاصل في كل من مستشفى المواساة ومستشفى الأسد الجامعي بدمشق بعد توقيعهم على الموافقة المستنيرة. حقق المرضى جميعهم المعايير التشخيصية التي وضعتها الكلية الأمريكية لطب الروماتيزمAmerican College of (ACR) Rheumatology عام 1987، بعد مشورة طبيب مفاصل أخصائي لتأكيد التشخيص (6).
جرى العمل في مخبر البيولوجيا الجزيئية بمستشفى الأسد الجامعي بدمشق. اشتملت عينة الدراسة على 13 عينة دنا DNA استُخلِصَت من ثلاثة عشر مريضاً بواسطة عتيدة high pure PCR template preparation kit من صنع شركة روش الألمانية، وأخضِعت إلى تفاعل سلسلة الدنا DNA sequencing لمعرفة النمط الجيني لـ STAT4 rs7574865. جرى استخدام 9/13 عينة من هذه العينات معروفة النمط الجيني في أمثلة optimization تفاعل ARMS-PCR وأخضعت بعدها العينات معروفة النمط الجيني جميعها إلى تفاعل الـ ARMS-PCR بعد أمثلته.
أمثلة تفاعل ARMS–PCR باستخدام عينات معروفة النمط الجيني
اعتُبِر النمط الجيني GG النمط الجيني الشائع والنمط الجيني GT النمط الجيني الطافر متغاير الألائل، في حين اعتُبِر النمط الجيني TT النمط الجيني الطافر متماثل الألائل.
انقسم العمل بشكل رئيسي إلى قسمين:
أولهما كان تقييماً لمشارع منشورة (ARMS G، ARMS T، ARMS C، S3، S4) في دراسات سابقة. وثانيهما جرى العمل فيه على تصميم مشارع جديدة وتقييمها ARMS G new)، ARMS T new). وجرى تصميم المشارع الجديدة باستخدام برمجية WASP وبرمجية Batch Primer 3(7، 8). كما جرى التحقق من اصطفاف المشارع المتبنّاة جميعها والمصممة مع تسلسل جين STAT4 في المنطقة المدروسة باستخدام NCBI nucleotide BLAST. صنعت جميع المشارع في شركة VBC Biotech Services (فيينا، النمسا) (الجدول 1).
التجربة الأولى: استخدم كل من المشرعين ARMS G وARMS T مع المشرع العكسي المشترك ARMS C في أنبوبين منفصلين لكل عينة. استخدمت في هذه المرحلة من التجارب تراكيز مختلفة من المشارع (0.1، 0.3، 0.5 مكرومول/ ل)، بالإضافة إلى درجات حرارة تلدين مختلفة (50، 51، 52، 53°C) (الجدول 2).
التجربة الثانية: استخدم المشرعان ARMS G وARMS T ولكن مع المشرع العكسي S4 في أنبوبين منفصلين لكل عينة بتركيز 0.5 مكرومول/ ل في كل أنبوب من هذه المشارع. وأجريت ثلاث تجارب باستخدام درجات حرارة تلدين مختلفة (53، 54، 55°C) (الجدول 2).
التجربة الثالثة: استخدم المشرعان المصممان ARMS G new، ARMS T new مع المشرع S3 بتراكيز مختلفة لهذه المشارع (0.1، 0.3، 0.5، 0.6، 0.7 مكرومول/ ل). استعملت في هذه المرحلة درجات حرارة تلدين مختلفة (60، 61، 61.3، 61.5، 62°C) (الجدول 2).
|
 |
| أُجريت تفاعلات الأمثلة السابقة جميعها باستخدام عتيدة HotStar PCR SuperMix kit (GeneDirex, Las Vegas, NV)، على جهاز (Eppendorf Hamburg Master Cycler® Pro S, Germany) لكل تفاعل تضخيم، وذلك بإتباع البرنامج الحراري المتضمن Hot start لمدة دقيقتين بدرجة حرارة C°94، ومن ثم تثبيت درجة حرارة C°94 (درجة حرارة تمسخ) لمدة 30 ثانية، و30 ثانية عند الوصول إلى درجات حرارة التلدين التجريبية، ثم 72C? (درجة حرارة إطالة) لمدة 30 ثانية. أنهيت التفاعلات بعد إتمام 40 دورة لكل منها دون اللجوء إلى عملية إطالة نهائية Final extension. |
| النتائج Results |
نتائج المرحلة الأولى من الأمثلة
عند استخدام عينة من النمط الشائع GG ظهرت العصابة 244 bp الناتجة عن تفاعل التضخيم عند إجراء الرحلان الكهربائي على الهلامة في جميع الأنابيب الحاوية على مشرع النمط الشائعARMS G ولكن ظهرت أيضاً مع مشرع النمط الطافر (ARMS T)، باستخدام درجة حرارة تلدين (50، 51، 52°C) مع تغيير تركيز المشرع (بين 0.3 و0.5 مكرومول/ ل). ولم تظهر أي عصابة عند استخدام تركيز 0.1 مكرومول/ ل من المشارع مع درجة حرارة تلدين 50°C. بالإضافة إلى ذلك، أبدت جميعها عصابات غير نوعية (الجدول 3).
نتائج المرحلة الثانية من الأمثلة
ظهرت العصابات 58 bp في جميع الأنابيب الحاوية على مشرع النمط الشائع ARMS G ومشرع النمط الطافر ARMS T عند استخدام درجة حرارة تلدين 53، 54°C. ولم تظهر أي عصابة في درجة حرارة تلدين C°55، واستمرت العصابات غير النوعية في الظهور (الجدول 4).
نتائج المرحلة الثالثة من التجارب
عند استخدام عينة من النمط الشائع GG ومشرع النمط الشائع المصمم ARMS G new بتراكيز 0.1، 0.3، 0.5، 0.6، 0.7 مكرومول/ ل بدرجة حرارة تلدين C°62، لم تظهر أي عصابة تضخيم،
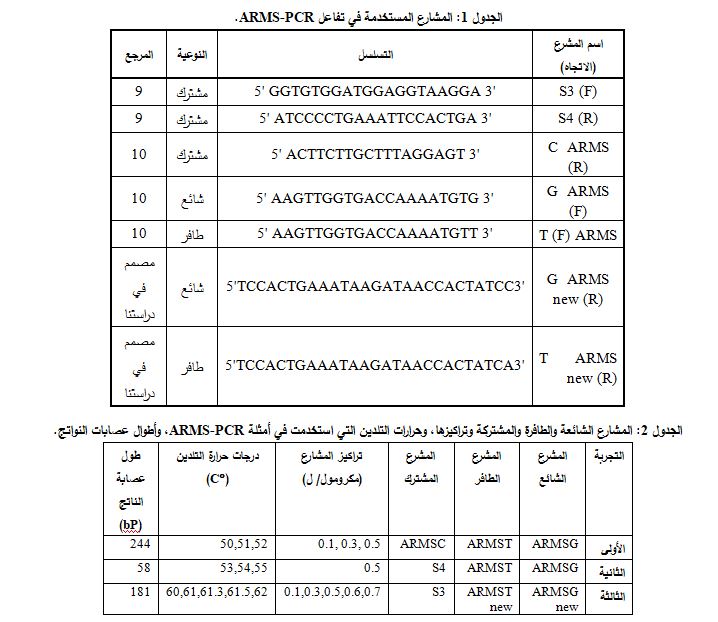
وكذلك لم تظهر أي عصابة عند استخدام مشرع النمط الطافر المصمم ARMS T new بالتراكيز 0.1، 0.3، 0.5، 0.6، 0.7 مكرومول/ ل وبدرجة حرارة تلدين C°62. وعند إجراء التفاعلات بالشروط المذكورة سابقاً ولكن باستخدام درجة حرارة تلدين C°60 وإجرائها أيضاً باستخدام درجة حرارة تلدين C°61، ظهرت عصابة بطول 181 bp في كلا حالتي مشرع النمط الشائع ومشرع النمط الطافر.
أظهرت نتائجنا أنه عند اتباع البرنامج الحراري المتضمن إما درجة حرارة التلدينC°61.3 أو درجة حرارة التلدين C°61.5، واستخدام أي من تراكيز مشرع النمط الشائع ظهرت عصابة بطول 181 bp، ولم تظهر أي عصابة باستخدام أي من تراكيز مشرع النمط الطافر (الجدول 5، الشكل 1).
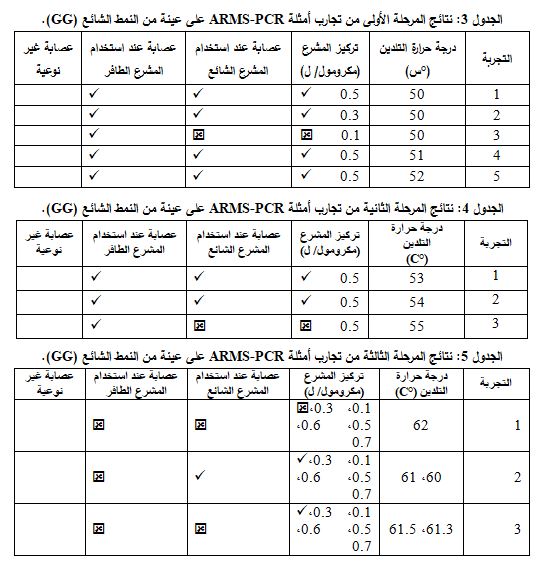
نتيجة لإجراءات الأمثلة تم اعتماد المشرع S3 كمشرع مشترك بتركيز مماثل لتركيز المشرع الآخر، والمشرع ARMS G new كمشرع للنمط الشائع بتركيز 0.3 مكرومول/ ل، والمشرع ARMS T new كمشرع للنمط الطافر بتركيز 0.5 مكرومول/ ل، والتضخيم باستخدام برنامج حراري يتضمن درجة حرارة تلدين C°61.5 لكلا المشرعين (الجدول 6).
|
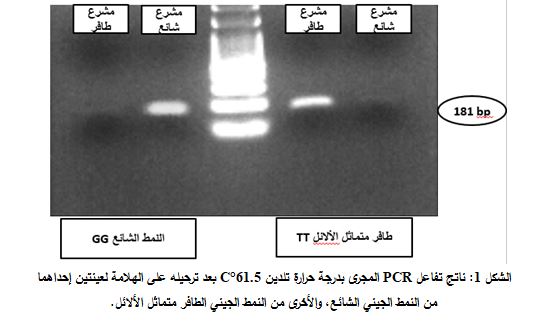 |
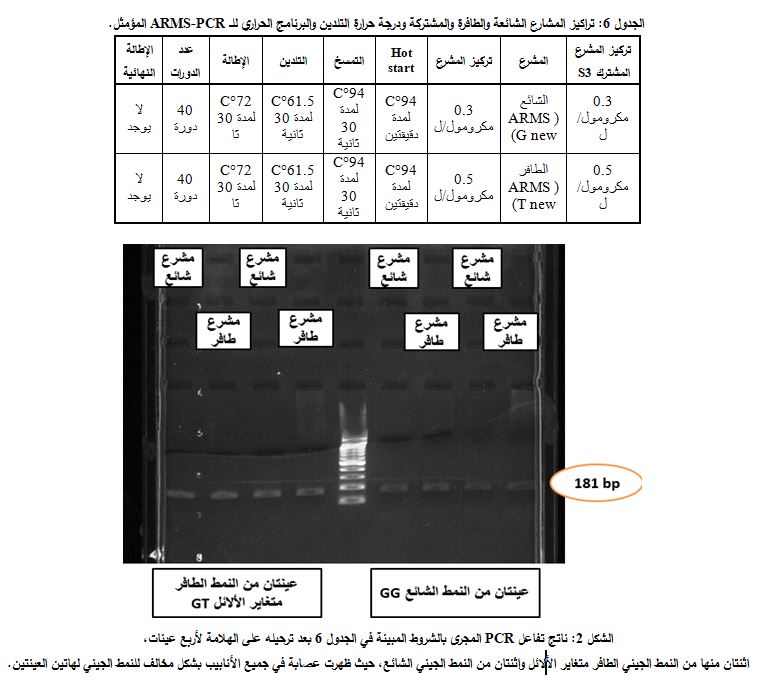
نتائج التنميط الجيني للعينات معروفة النمط باستخدام ARMS-PCR المؤمثل
عند تطبيق تفاعل ARMS-PCR بشروطه المبينة في الجدول 6 على العينات المدروسة، أظهرت عينتان (2/13) من هذه العينات نتيجة مغايرة لنتيجة تفاعل السلسلة. حيث أبدت هاتان العينتان عصابة في ناتج رحلان كلا أنبوبي التفاعل، مما يدل على أن النمط الجيني متغاير الألائل GT، في حين أنها كانت بنمطGG عندما جرى تنميطها باستخدام تفاعل السلسلة (الشكل 2).
|
 |
| المناقشة Discussion |
توصلت دراستنا إلى الشروط الحرارية وتركيز المشارع لإجراء تفاعل ARMS-PCR من أجل التنميط الجيني للتعدد الشكلي STAT4 rs7574865.
تبين أن المشارع المنشورة في دراسات سابقة (9، 10) والمستخدمة في دراستنا تتمتع بدرجة حرارة انصهار منخفضة (ARMS C Tm=49.1°)، كما أوضحت الشركة المصنعة لها. وهذا اضطرنا إلى استخدام درجات حرارة تلدين قريبة منها (50، 51، 52C°). وهذا يفسر لا نوعية التفاعلات وظهور العصابات غير النوعية. كما أظهرت نتائجنا أنه حتى عند استثناء المشرع ARMS C ذي درجة حرارة الانصهار المنخفضة ورفع درجة حرارة التلدين إلى 53، 54، 55°C، لم نحصل على تفاعلات نوعية وظهرت العصابات غير النوعية، وهذا قد يكون مرده إلى درجات حرارة الانصهار لكل من المشرعين النوعيين ARMS G وARMS T (51، 53°C)، على الترتيب، المنشورين في الدراسات السابقة. علماً بأنه يُنصح بأن تكون الدرجة C°60 فما فوق هي درجة حرارة التلدين لتعطي نوعية أفضل عند إتباع طريقة ARMS-PCR (11). إثر الحصول على النتائج غير النوعية باستخدام المشارع المنشورة في الدراسات السابقة (ARMS G, ARMS T, ARMS C) قمنا بتصميم مشارع خاصة بدراستنا. حيث راعينا في هذا التصميم أن تكون بطول27 نوكليوتيداً، بينما كان طول تلك المذكورة في الدراسات 20 نوكليوتيد. بالإضافة إلى تضمين تصميمنا نوكليوتيد إضافي غير مطابقadditional mismatch في كلا المشرعين النوعيين المصممين (ARMS G new, ARMS T new) بهدف زيادة النوعية. وجرى إدخال الـ mismatch بإتباع القواعد التي أرساها Little وبالاستعانة بالبرمجيات الحاسوبية (7، 12).
ما أوضحته نتائجنا من عدم تطابق نتائج التنميط الجيني للتعدد الشكلي STAT4 rs7574865 باتباع طريقتي السلسلة sequencing وARMS-PCR، بالرغم من اتباع الشروط المثالية التي حصلنا عليها، يمكن أن يعزى إلى دور شاردة المغنزيوم Mg2+ كعامل مساعد لعمل البوليميراز (11) الذي لم نتمكن من دراسة تراكيز مختلفة منها، كونها موجودة بتركيز محدد في مزيج التفاعل ضمن العتيدة المستخدمة.
كما يجب التنويه إلى أن أمثلة تفاعل ARMS-PCR في بعض الحالات تكون أكثر صعوبة منها في حالات أخرى وذلك حسب نوع الطفرة. لما كان التعدد الشكلي STAT4 rs7574865من النوع G > T، فإن عدم التطابق بين المشرع والمرصاف الطافر سيكون من النوع C / T والذي سيؤدي إلى زعزعة ضعيفة للثباتية والاستقرار weak Destabilizing effect (1). بينما تذكر دراسة Little بأن للـ C / T أثراً مزعزعاً قوياً عائداً إلى صعوبة تشكل الروابط الهيدروجينية بين نوكليوتيدين من النوع بريميدين – بريميدين (12).
الاستنتاجات
نخلص من دراستنا إلى أن أمثلة تفاعل ARMS-PCR تتأثر بشكل رئيسي بدرجة حرارة التلدين وتركيز المشارع المستخدمة، بالإضافة إلى الدور المحتمل لشاردة المغنزيوم، وتتطلب دراسة شاملة ودقيقة لنوع الطفرة، وعدم التطابق الحاصل، وتصميم المشارع المستخدمة، حيث أنها طريقة سهلة ممكنة التطبيق ورخيصة ولا تحتاج إلى أجهزة معقدة.
|
| المراجع References |
1- Liu J. et al.
An improved allele specific PCR primer design method for SNP marker analysis and its application.
Plant Methods. 2012 Aug 24; 8(1):34. doi: 10.1186/1746-4811-8-34.
2- Gaudet M. et al.
Allele-Specific PCR in SNP Genotyping. In: Komar A editor. Single Nucleotide Polymorphisms-Methods and Protocols.
2nd ed. Humana press; 2012. P 415-425.
3- Tong G. et al.
Association between polymorphism in STAT4 gene and risk of rheumatoid arthritis: a meta-analysis.
Hum Immunol. 2013 May; 74(5):586-92. doi: 10.1016/ j. humimm. 2012.11.033. Epub 2013 Jan 19.
4- Shen L. et al.
Replication study of STAT4 rs7574865 G/T polymorphism and risk of rheumatoid arthritis in a Chinese population.
Gene. 2013 Sep 10; 526(2): 259-64. doi: 10. 1016/j. gene. 2013.05.022. Epub 2013 May 30.
5- Lamana A. et al.
The Minor Allele of rs7574865 in the
STAT4 Gene Is Associated with Increased mRNA and ProteinExpression. PLoS One. 2015 Nov 16;10(11): e0142683. doi: 10.1371
Journal pone. 0142683. e Collection 2015.
6- O’Dell JR et al.
Rheumatoid Arthritis. In: John B. Imboden editor. Rheumatology current diagnosis and treatment 3rd ed. McGraw-Hill Education; 2013. p 139- 150.
7- Wangkumhang P. et al.
WASP: a Web-based Allele-Specific PCR assay designing tool for detecting SNPs and mutations.
BMC Genomics. 2007 Aug 14;8:275.
8- Frank M. et al.
Batch Primer 3: a high throughput web application for PCR and sequencing primer design.
BMC Bioinformatics, 2008, 9: 253, doi: 10.1186/1471-2105-9-253
9- Su Y et al.
Variation in STAT4 is associated wih systemic lupus erythematosus in Chinese Northern Han population.
Chin Med J (Engl). 2010 Nov; 123(22): 3173-3177.
10- ZHU H. et al.
Polymorphisms of STAT4 and the risk of inflammatory bowel disease: A case-control study in Chinese Han population.
Biomed Rep. 2013 Mar; 1(2): 320-324. Epub 2013 Jan 21.
11- Medrano RF et al.
Guidelines for the tetra-primer ARMS-PCR technique development.
Mol Biotechnol. 2014 Jul; 56(7): 599-608. doi: 10.1007/s12033-014-9734-4.
12- Little S.
Amplification-Refractory Mutation System (ARMS) Analysis of Point Mutations.
Curr. Protoc. Hum. Genet. 2001; 7: 9. 8. 1–9. 8. 12.
|
| |
